Recâblage progressif de la chromatine par ETO2::GLIS2 révélé dans un modèle iPSC humain à génome modifié d’initiation de leucémie pédiatrique
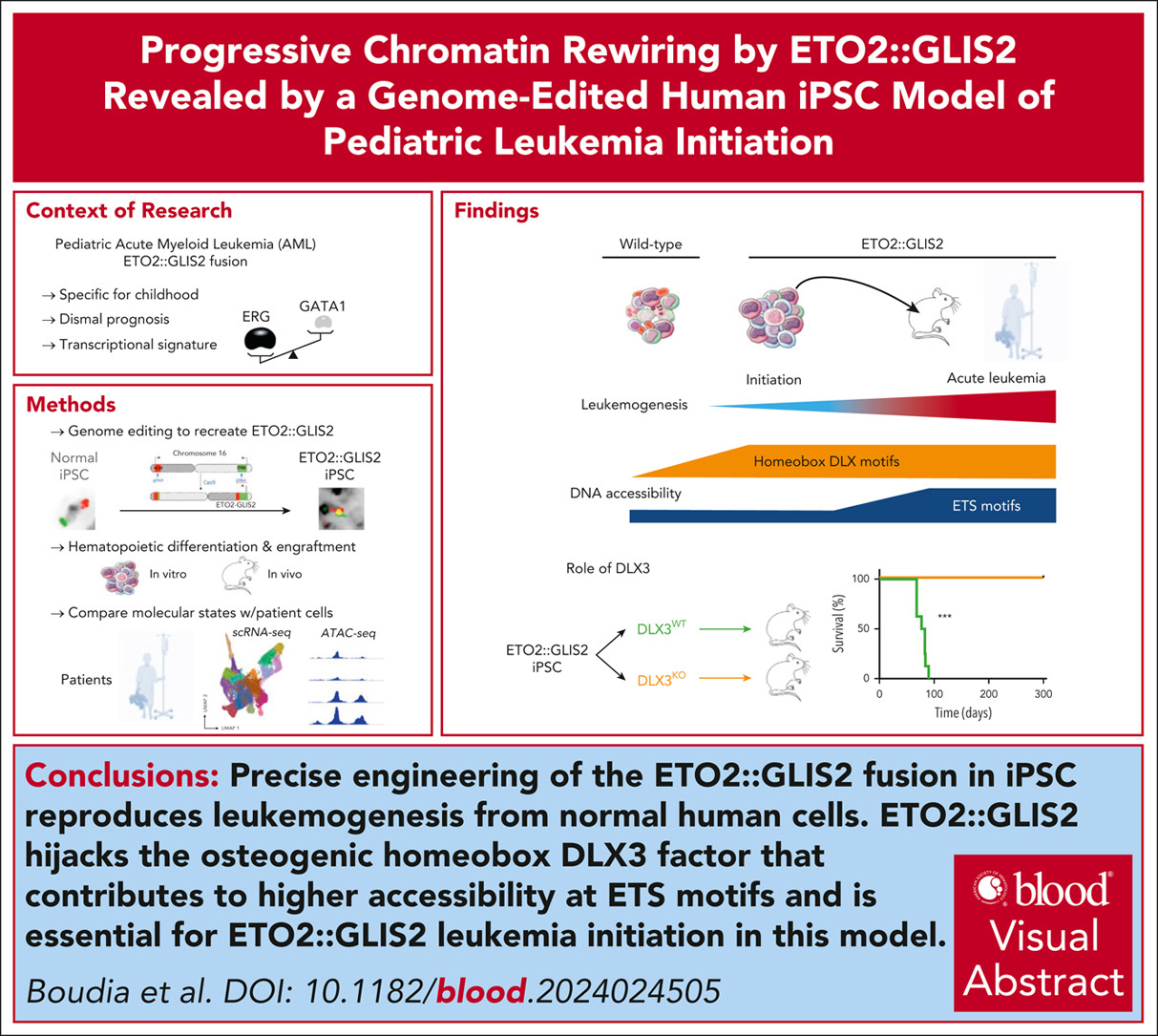
La leucémie myéloïde aiguë pédiatrique héberge fréquemment des oncogènes de fusion associés à un mauvais pronostic, notamment des réarrangements KMT2A, NUP98 et GLIS2. Bien que des modèles murins aient démontré leurs activités leucémiques, les étapes d’une cellule humaine normale aux blastes leucémiques restent incertaines. Ici, nous avons reproduit avec précision l’inversion du chromosome 16 aboutissant à la fusion d’ETO2 ::GLIS2 dans des cellules souches pluripotentes induites humaines (iPSC).
Les cellules hématopoïétiques exprimant ETO2 ::GLIS2 dérivées d’IPSC ont montré des altérations de différenciation in vitro et ont efficacement induit le développement in vivo d’une leucémie qui a étroitement phénocopié la leucémie mégacaryoblastique aiguë humaine (AMKL) reflétée par la cytométrie en flux et les transcriptomes de cellules uniques.
La comparaison des cellules dérivées d’iPS avec des cellules dérivées de patients a révélé une altération de l’accessibilité de la chromatine aux stades précoces et ultérieurs de la leucémie de bonne foi, avec une accessibilité et une expression plus élevées et aberrantes du facteur homéogène DLX3 qui ont précédé une accessibilité accrue aux facteurs ETS. La surexpression de DLX3 dans les cellules CD34 normales a augmenté l’accessibilité aux motifs ETS et réduit l’accessibilité aux motifs GATA.
Un module transcriptionnel DLX3 a été globalement enrichi à la fois en ETO2 ::GLIS2 AMKL et en un ostéosarcome pédiatrique agressif. Il est important de noter que l’inactivation de DLX3 a abrogé l’initiation de la leucémie dans ce modèle iPSC ETO2 ::GLIS2. Collectivement, la caractérisation d’un nouveau modèle AMKL humain dérivé d’iPSC a révélé que le détournement du facteur de transcription ostéogénique homéobox DLX3 était une étape précoce essentielle dans les changements de la chromatine et la leucémogenèse induits par l’oncogène de fusion ETO2 ::GLIS2.